通常通过协方差矩阵的特征分解来解释主成分分析(PCA)。但是,它也可以通过数据矩阵奇异值分解(SVD)来执行。它是如何工作的?这两种方法之间有什么联系?SVD和PCA之间是什么关系?
换句话说,如何使用数据矩阵的SVD进行降维?
通常通过协方差矩阵的特征分解来解释主成分分析(PCA)。但是,它也可以通过数据矩阵奇异值分解(SVD)来执行。它是如何工作的?这两种方法之间有什么联系?SVD和PCA之间是什么关系?
换句话说,如何使用数据矩阵的SVD进行降维?
Answers:
令数据矩阵为大小,其中是样本数,是变量数。让我们假设它是居中的,即列均值已经减去,现在等于零。 Ñ × p Ñ p
然后协方差矩阵由。它是一个对称矩阵,因此可以对角线化:其中是特征向量的矩阵(每列是一个特征向量),而是对角矩阵,其特征值在对角线上递减。特征向量被称为数据的主轴或主轴方向。数据在主轴上的投影称为主成分,也称为PC分数C ^ C ^ = X ⊤ X /(ñ - 1 )C ^ = V 大号V ⊤,V 大号λ 我 Ĵ Ĵ X V我我X V
如果现在执行奇异值分解,我们将获得分解其中是ary矩阵,是对角矩阵奇异值。从这里可以轻松地看到表示右奇异矢量是主要方向,并且奇异值通过与协方差矩阵的特征值相关。主要成分由X = û 小号V ⊤,û 小号小号我Ç = V 小号ü ⊤ ù 小号V ⊤ /(ñ - 1 )= V 小号 2
总结一下:
SVD和PCA之间的直观关系是什么 -Math.SE上非常流行且非常相似的线程。
为什么通过数据SVD进行数据PCA?-讨论通过SVD执行PCA的好处[简短答案:数值稳定性]。
PCA和对应关系分析与Biplot的关系 -在某些同类技术的背景下,PCA均基于SVD。
与PCA相比,SVD有什么优势吗?-一个问题,询问使用SVD 代替 PCA 是否有任何好处[简短答案:不适定问题]。
理解主成分分析,特征向量和特征值 -我的回答给出了PCA的非技术性解释。为了引起注意,我在这里复制一个图:
我写了一个Python&Numpy片段,附有@amoeba的答案,如果对某人有用,我将其留在此处。这些评论大部分来自@amoeba的答案。
import numpy as np
from numpy import linalg as la
np.random.seed(42)
def flip_signs(A, B):
"""
utility function for resolving the sign ambiguity in SVD
http://stats.stackexchange.com/q/34396/115202
"""
signs = np.sign(A) * np.sign(B)
return A, B * signs
# Let the data matrix X be of n x p size,
# where n is the number of samples and p is the number of variables
n, p = 5, 3
X = np.random.rand(n, p)
# Let us assume that it is centered
X -= np.mean(X, axis=0)
# the p x p covariance matrix
C = np.cov(X, rowvar=False)
print "C = \n", C
# C is a symmetric matrix and so it can be diagonalized:
l, principal_axes = la.eig(C)
# sort results wrt. eigenvalues
idx = l.argsort()[::-1]
l, principal_axes = l[idx], principal_axes[:, idx]
# the eigenvalues in decreasing order
print "l = \n", l
# a matrix of eigenvectors (each column is an eigenvector)
print "V = \n", principal_axes
# projections of X on the principal axes are called principal components
principal_components = X.dot(principal_axes)
print "Y = \n", principal_components
# we now perform singular value decomposition of X
# "economy size" (or "thin") SVD
U, s, Vt = la.svd(X, full_matrices=False)
V = Vt.T
S = np.diag(s)
# 1) then columns of V are principal directions/axes.
assert np.allclose(*flip_signs(V, principal_axes))
# 2) columns of US are principal components
assert np.allclose(*flip_signs(U.dot(S), principal_components))
# 3) singular values are related to the eigenvalues of covariance matrix
assert np.allclose((s ** 2) / (n - 1), l)
# 8) dimensionality reduction
k = 2
PC_k = principal_components[:, 0:k]
US_k = U[:, 0:k].dot(S[0:k, 0:k])
assert np.allclose(*flip_signs(PC_k, US_k))
# 10) we used "economy size" (or "thin") SVD
assert U.shape == (n, p)
assert S.shape == (p, p)
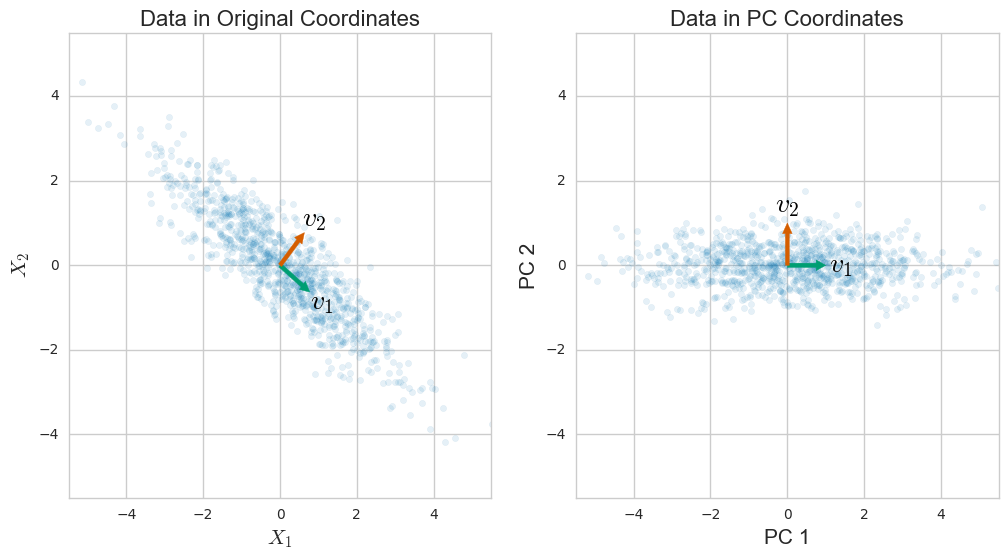
assert V.shape == (p, p)让我从PCA开始。假设您有n个数据点,每个数据点包含d个数字(或维)。如果将此数据居中(从每个数据向量减去平均数据点),则可以堆叠数据以形成矩阵
协方差矩阵
度量在不同程度上给出数据的不同坐标。因此,PCA(旨在捕获数据的变化)可以根据协方差矩阵给出,这并不奇怪。特别地,该特征值分解原来是
其中是第个主成分,即PC,是第个特征值,并且还等于第个PC上数据的方差。这种分解来自线性代数中的一个一般性定理,并且确实需要做一些工作来促使relatino转向PCA。我λ 我我小号我
SVD是一种根据矩阵的列空间和行空间来理解矩阵的一般方法。(这是一种以与行和列空间具有直观关系的其他矩阵形式重写任何矩阵的方法。)例如,对于矩阵我们可以在域和范围内找到和方向,以便 u i v i
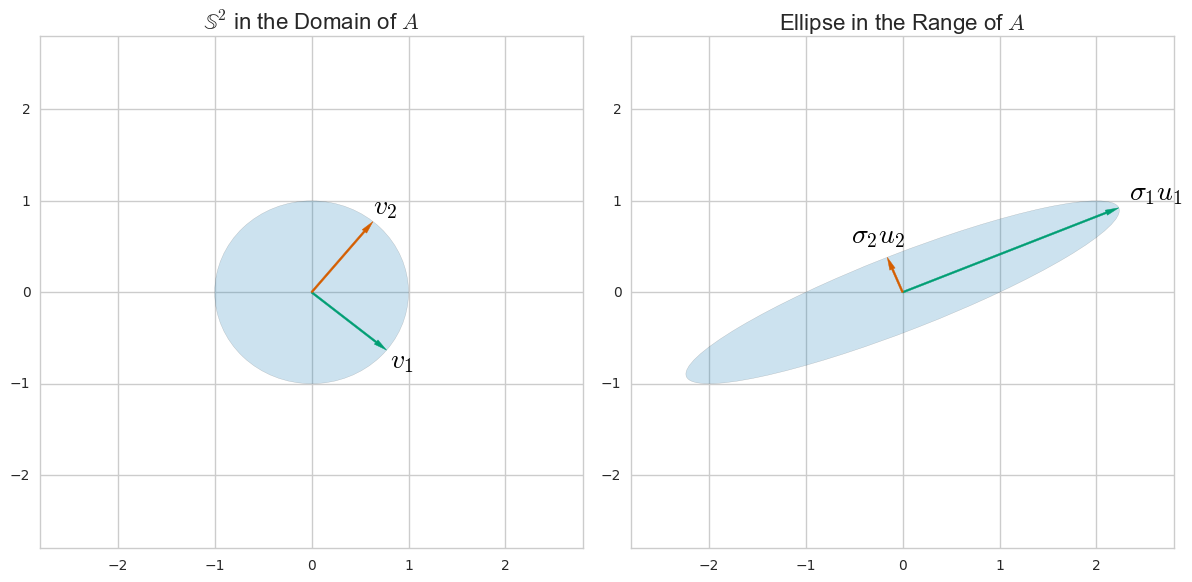
和“奇异值”以下方式与数据矩阵相关
通常的事实是,右奇异向量跨越的列空间。在这种特定情况下,给我们数据在第个主分量方向上的缩放投影。左奇异向量通常跨越的行空间,这给了我们一组正交向量,它们像PC一样跨数据。 X ü 我 X 我v 我 X
在这篇较长的文章中,我将探讨PCA和SVD之间关系的更多细节和好处。