将数据居中(或取消定义)对PCA有什么区别?我听说它可以简化数学运算,也可以防止第一台PC被变量的均值控制,但是我觉得我还不能完全掌握这个概念。
例如,此处的最佳答案是如何使数据居中摆脱回归和PCA中的截距?描述了不居中如何将第一个PCA拉过原点,而不是点云的主轴。基于我对如何从协方差矩阵的特征向量中获得PC的理解,我不明白为什么会发生这种情况。
而且,我自己进行的有无居中的计算似乎毫无意义。
考虑irisR 中的数据集中的setosa花。我按如下方式计算了样本协方差矩阵的特征向量和特征值。
data(iris)
df <- iris[iris$Species=='setosa',1:4]
e <- eigen(cov(df))
> e
$values
[1] 0.236455690 0.036918732 0.026796399 0.009033261
$vectors
[,1] [,2] [,3] [,4]
[1,] -0.66907840 0.5978840 0.4399628 -0.03607712
[2,] -0.73414783 -0.6206734 -0.2746075 -0.01955027
[3,] -0.09654390 0.4900556 -0.8324495 -0.23990129
[4,] -0.06356359 0.1309379 -0.1950675 0.96992969如果我首先对数据集进行居中,则会得到完全相同的结果。这似乎很明显,因为居中根本不会改变协方差矩阵。
df.centered <- scale(df,scale=F,center=T)
e.centered<- eigen(cov(df.centered))
e.centeredprcomp对于居中和未居中的数据集,该函数也会精确生成此特征值-特征向量组合。
p<-prcomp(df)
p.centered <- prcomp(df.centered)
Standard deviations:
[1] 0.48626710 0.19214248 0.16369606 0.09504347
Rotation:
PC1 PC2 PC3 PC4
Sepal.Length -0.66907840 0.5978840 0.4399628 -0.03607712
Sepal.Width -0.73414783 -0.6206734 -0.2746075 -0.01955027
Petal.Length -0.09654390 0.4900556 -0.8324495 -0.23990129
Petal.Width -0.06356359 0.1309379 -0.1950675 0.96992969但是,该prcomp功能具有默认选项center = TRUE。禁用此选项将导致以下PC的未居中数据(设置为false p.centered时保持不变center):
p.uncentered <- prcomp(df,center=F)
> p.uncentered
Standard deviations:
[1] 6.32674700 0.22455945 0.16369617 0.09766703
Rotation:
PC1 PC2 PC3 PC4
Sepal.Length -0.8010073 0.40303704 0.4410167 0.03811461
Sepal.Width -0.5498408 -0.78739486 -0.2753323 -0.04331888
Petal.Length -0.2334487 0.46456598 -0.8317440 -0.19463332
Petal.Width -0.0395488 0.04182015 -0.1946750 0.97917752为什么这与我自己对无中心数据的协方差矩阵进行的特征向量计算不同?它与计算有关吗?我已经看到提到过prcomp使用SVD方法而不是特征值分解来计算PC的方法。该函数princomp使用后者,但其结果与相同prcomp。我的问题与我在本文顶部描述的答案有关吗?
through the origin, rather than the main axis of the point cloud。PCA总是刺破起源。如果数据居中,则原点=重心。
请参阅“ 主成分分析:综述和最新动态”,以讨论中心PCA和非中心PCA(通常是不同的)。
—
杨艺博
















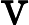













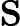



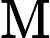




Based on my understanding of how the PC's are obtained from the covariance matrix's eigenvectors...请阅读您链接到的答案中的注释。协方差表示数据居中,“协方差”上的PCA =中心数据上的PCA。如果不将原始变量居中X,则基于此类数据的PCA将为X'X/n [or n-1]矩阵上的= PCA 。另请参阅重要概述:stats.stackexchange.com/a/22520/3277。